QOJ.ac
QOJ
# 10182. 진화 2
Statistics我们正在研究生物的进化历程。除了最初的生物外,所有生物都由已存在的生物进化而来。我们把已存在的生物称为父母生物,新诞生的生物称为子生物。
N 个生物各有一个独一无二的出生编号,范围从 0 到 N−1。这些编号按生物诞生的顺序分配,因此父母生物的出生编号总是小于子生物的编号,而最初生物的出生编号为 0。
生物通过进化诞生的过程可以用一个树形结构来表示:生物是树中的节点,进化关系是连接父母生物和子生物的边,最初生物作为树的根。这个结构被称为进化树。
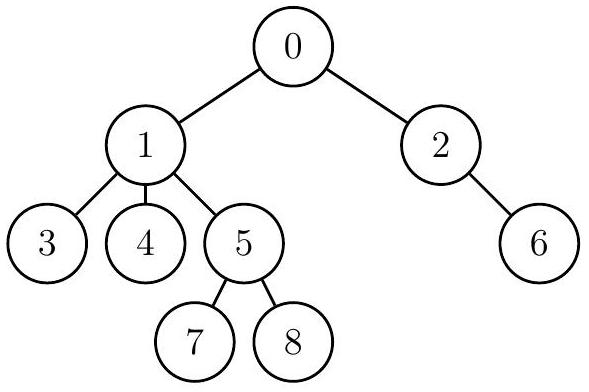
例如,假设最初编号为 0 的生物进化出编号为 1 和 2 的生物,编号 1 的生物进化出编号 3、4、5 的生物,编号 2 的生物进化出编号 6 的生物,而编号 5 的生物又进化出编号 7 和 8 的生物,这样的进化树如下图所示,每个节点标有生物的出生编号。
但不幸的是,你的珍贵进化树不小心洒上了咖啡。咖啡浸湿后,进化树的形状和最初生物(编号 0)还能辨认,但除最初生物外的其他生物的出生编号已无法识别。
你赶紧给每个生物临时编了个编号:最初生物的临时编号仍为 0,其余 N−1 个生物被随机分配了 1 到 N−1 的不同编号。这些临时编号可能与出生编号相同,也可能不同,且不像出生编号那样保证父母生物的编号小于子生物。
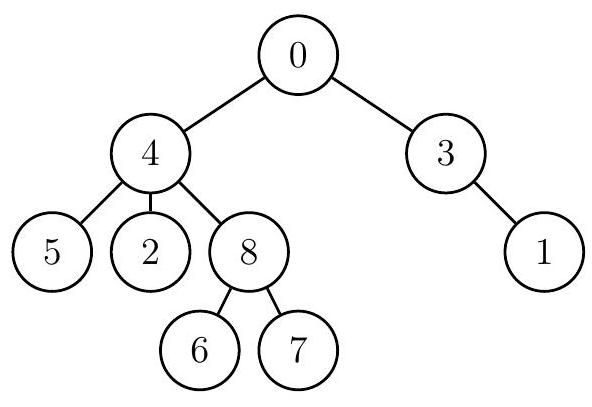
例如,同一进化树可能被临时编号如下图所示,每个节点标有临时编号。
幸运的是,你的电脑里存有进化树的备份,但备份文件被加密,你忘记了密码。好在备份程序还能用。程序提供了一个功能:输入两个生物的临时编号,它会告诉你哪个生物的出生编号更小。但若频繁使用这个功能,你的电脑可能会崩溃。因此,你需要编写代码,用尽量少的查询次数恢复进化树中所有生物的出生编号。
实现细节
你需要实现以下函数:
std::vector<int> recover(int N, std::vector<int> U, std::vector<int> V)- 该函数在一次执行中可能被调用多次。
U, V:长度为 N−1 的整数数组。对于任意 0≤i≤N−2,表示进化树中临时编号为 U[i] 的生物是父母,临时编号为 V[i] 的生物是子生物。所有 V[i] 互不相同。- 你需要在每次调用中,通过调用后续定义的
compare函数(可调用 0 次或多次),找出进化树中每个生物的出生编号,并将其存入一个大小为 N 的std::vector返回。设返回值为 X,则对于所有 0≤i≤N−1,临时编号为 i 的生物的出生编号应为 X[i]。
程序可调用以下函数:
int compare(int a, int b)- a 和 b 必须满足 0≤a,b≤N−1 且 a≠b。
- 若临时编号为 a 的生物的出生编号小于临时编号为 b 的生物的出生编号,返回 1;否则返回 0。
你提交的代码不得包含任何输入输出函数。
样例数据
假设 N=4,U=[0,0,0],V=[1,2,3]。评测程序调用:
recover(4, [0, 0, 0], [1, 2, 3])树的结构如下图:
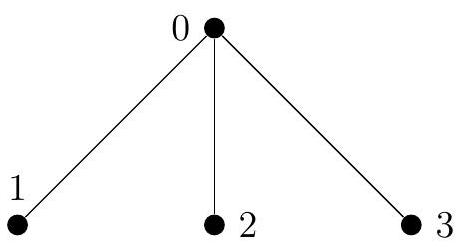
假设出生编号如下: - 临时编号 0:出生编号 0(固定) - 临时编号 1:出生编号 2 - 临时编号 2:出生编号 3 - 临时编号 3:出生编号 1
初始时,可能的出生编号排列有 [0,1,2,3],[0,1,3,2],[0,2,1,3],[0,2,3,1],[0,3,1,2],[0,3,2,1],共 P=6,故 Z=⌈log26⌉=3。
通过以下调用:
- compare(1, 2) 返回 1(2<3)
- compare(2, 3) 返回 0(3>1)
- compare(1, 3) 返回 0(2>1)
- compare(0, 3) 返回 1(0<1)
调用 4 次后返回 [0,2,3,1],结果正确。此时 C=4,K=43≤1.4,得满分。此样例满足子任务 2 和 4 的条件。
子任务
对于所有输入数据,满足:
- 2≤N≤10000
- 对于所有 0≤i≤N−2:
- 0≤U[i]≤N−1
- 1≤V[i]≤N−1
- U[i]≠V[i]
- 输入数据构成一个有效的进化树。
- 一次执行中所有
recover调用中 N 的总和不超过 10000。 - 本题的评测程序是非自适应的(NOT adaptive),即各生物的出生编号在程序开始时已固定,不会因
compare调用而改变。
详细子任务附加限制及分值如下表所示。
| 子任务 | 分值 | 附加限制 |
|---|---|---|
| 1 | 1 | 进化树中不存在与三个以上生物相邻的生物,且最初生物只与一个生物相邻 |
| 2 | 7 | 对于所有 0≤i≤N−2,U[i]=0 |
| 3 | 12 | 可为进化树中的每个生物重新分配一个颜色 c (0≤c≤N−1),使得对于所有 1≤i≤N−1,颜色为 i 的生物的父母颜色为 ⌊i−12⌋,且存在正整数 k 满足 N=2k−1。即进化树是完全二叉树 |
| 4 | 80 | 无附加限制 |
每次 recover 调用按以下方式评分。各子任务的得分取该子任务内所有测试用例中 recover 调用得分的最小值:
- 若程序异常终止或
recover返回值错误,得 0 分。 在未知
compare提供的编号大小关系及子任务限制时,对于 0≤i≤N−1,临时编号为 i 的生物的出生编号为 X[i] 的可能排列 X(0,1,⋯,N−1 的顺序)数量记为 P。因正确答案也是可能的 X 之一,P 为正整数。定义 Z=⌈log2P⌉,C 为本次调用中compare的调用次数。得分由 K=CZ 决定。若 Z=0 导致 K 未定义,则当 C=0 时 K=0,当 C>0 时 K=2025。若 K>20,得 0 分。
- 若 8<K≤20,得该子任务分数的 (5×20−K12+5)%。
- 若 2.5<K≤8,得该子任务分数的 (50×8−K5.5+10)%。
- 若 1.5<K≤2.5,得该子任务分数的 (20×(2.5−K)+60)%。
- 若 1.4<K≤1.5,得该子任务分数的 (10×1.5−K0.1+80)%。
- 若 K≤1.4,得该子任务分数的 100%。
样例交互库
示例评测程序接收测试用例数量 T,随后接收 T 组输入,每组包括:
- 第 1 行:N
- 第 2 行:PAR[1] PAR[2] ⋯ PAR[N−1]
- 第 3 行:Y[0] Y[1] ⋯ Y[N−1]
其中: - PAR[i]:出生编号为 i 的生物的父母出生编号,满足 0≤PAR[i]<i。 - Y[i]:出生编号为 i 的生物的临时编号,Y[0]=0,且 Y 是 0,1,⋯,N−1 的排列。
对每个测试用例,程序根据进化树构造 U 和 V,调用 recover,并输出:
- 若
compare(a, b)的 a,b 不满足 0≤a,b≤N−1,输出Wrong Answer [1]。 - 若 a=b,输出
Wrong Answer [2]。 - 若
recover返回数组长度不为 N,输出Wrong Answer [3]。 - 否则,第一行输出
C: 4(C 为compare调用次数),第二行输出recover返回值 X 的元素。
输出 Wrong Answer 后程序立即终止。
注意,正确输出应满足 Y[X[i]]=i,但示例程序不验证这一点,且可能与实际评测程序不同。